| Information of forward sequence (DDBJ: AG403964) |
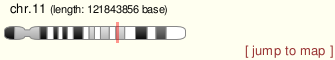
| Chromosome |
11 |
position(base) |
75237260 - 75237933 |
strand |
+ |
 |
| Alignment |
seq1: chr11_75237260_75237933
seq2: MSMg01-261K04.T7_53_717
seq1 ATTCATCAGTTTCCTATACCGCTGACTACACAGGGGCTGAAGGTAATCTC 50
|||||||||||| |||||||||||||||| ||||||||||||||||||||
seq2 ATTCATCAGTTTTCTATACCGCTGACTAC-CAGGGGCTGAAGGTAATCTC 49
seq1 AATGTTTTCATAACTGGTGTGGTGCTACTTGCTCATGATCCCAACACTTG 100
||||||||||||||||||||||||||||||||||||||||||||||||||
seq2 AATGTTTTCATAACTGGTGTGGTGCTACTTGCTCATGATCCCAACACTTG 99
seq1 GAAGGTAGGTCAGAAGTTCAAGAGCAGTCTTGACTACTCAGTGAATTTGA 150
||||||||||||||||||||||||||| ||||||||||||||||||||||
seq2 GAAGGTAGGTCAGAAGTTCAAGAGCAGCCTTGACTACTCAGTGAATTTGA 149
seq1 GGCTAGCCTGGGCTACATGAAAACTCATAAAACAATAAAAGAAAAGAAAA 200
|||||||||||||||||||||||||||||||||||| |||||||||||||
seq2 GGCTAGCCTGGGCTACATGAAAACTCATAAAACAATGAAAGAAAAGAAAA 199
seq1 GGTGGCGAGTGAGGTGGCCCATCAGGTAAAAGTGCCAACCACCTCGCCTG 250
||||||||||||||||||||||||||||||||||||||||||||||||||
seq2 GGTGGCGAGTGAGGTGGCCCATCAGGTAAAAGTGCCAACCACCTCGCCTG 249
seq1 AAAGCCTCCACACGGAAGGGGAAAGCCAGCTTCTACATGTGGTCCTCTGC 300
||||||||||||||||||| |||||||||||||||||||||||||||||
seq2 AAAGCCTCCACACGGAAGGTTAAAGCCAGCTTCTACATGTGGTCCTCTGC 299
seq1 CCTCCGCATGCACCATGGCTCGTGCACCCCCACACCCACCCACCCACCCA 350
||||||| |||||||||||||||||||||||| ||||||||||
seq2 CCTCCGCGTGCACCATGGCTCGTGCACCCCCA--------CACCCACCCA 341
seq1 CCCACATGACATAAATACTTGTAATGATTAGTTTCTGAAGAACAATATTT 400
||||||||||||||||||||||||||||||||||||||||||||||||||
seq2 CCCACATGACATAAATACTTGTAATGATTAGTTTCTGAAGAACAATATTT 391
seq1 TCGTTGATCTTGTTTAGGGAACAAAGTTCGTGCACATTGACCTGTCGACC 450
||||||||||||||||||||||||||||||||||||||||||||||||||
seq2 TCGTTGATCTTGTTTAGGGAACAAAGTTCGTGCACATTGACCTGTCGACC 441
seq1 GTGTAGTACGGGATCCGCTCCAGGAAGCTAAAGATTTTGGGATGCTTCAT 500
|||||||||||||||||||||| |||||||||||||||||||||||||||
seq2 GTGTAGTACGGGATCCGCTCCACGAAGCTAAAGATTTTGGGATGCTTCAT 491
seq1 GTTGCAGCTACTCTGATGAACAGTGTTG-CTTTCTGGGCCAGGTAATGGT 549
|||||||||||||||| |||||| | || ||||||||| |||||||||||
seq2 GTTGCAGCTACTCTGACGAACAGCGCTGCCTTTCTGGGTCAGGTAATGGT 541
seq1 GGCATATACCTTTGATCCCAGCACTTGGGAGGCAGAAGCATGTAGATCTC 599
||||||||||||||||||||||||||||||| |||||||||| |||||
seq2 GGCATATACCTTTGATCCCAGCACTTGGGAGCGCGAAGCATGTAAATCTC 591
seq1 TGTGAGTTCGAGATCAGCCTGGTCTACAGAGTGAGTTCCAGGATATCCAA 649
|||| |||||||| ||||||||| ||| |||| | || || ||||
seq2 TGTGTATTCGAGATTAGCCTGGTCCACATAGTGTAGCCAAGAATTTCCAG 641
seq1 GGCTATACAGAAAAACCCCTGTCTC 674
| |||| || || |||||||||||
seq2 GTCTATCCA-AATTACCCCTGTCTC 665
|
|
| Information of reverse sequence (DDBJ: AG403965) |
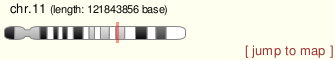
| Chromosome |
11 |
position(base) |
75322128 - 75322694 |
strand |
- |
 |
| Alignment |
seq1: chr11_75322128_75322694
seq2: MSMg01-261K04.TJ_69_637 (reverse)
seq1 TGGGATGGGGAGAAGACTATTATCATCACGTGCAGGTTGGTGTGGGCCTC 50
||||||||| ||||||||||||||||||| ||||||||||||||||||||
seq2 TGGGATGGGAAGAAGACTATTATCATCACATGCAGGTTGGTGTGGGCCTC 50
seq1 TTGGGTGGAAATATTATAGAGCAGGCATTGCAGGCCAGG-ACCTGGATTG 99
|||||||||||||||||||||||| ||||||||| |||| ||||||||||
seq2 TTGGGTGGAAATATTATAGAGCAGACATTGCAGGACAGGAACCTGGATTG 100
seq1 GTGGCTTGAGCGCTTACCCTGTCCCTGTCCCCCCCACCCCAGCTTCACAC 149
||||||||||||||||| |||| ||||| |||||||||||||||||||||
seq2 GTGGCTTGAGCGCTTACTCTGTACCTGTACCCCCCACCCCAGCTTCACAC 150
seq1 CAGGCTCCTGCACACTGACAGCCTACAAGCTGACACCCAGTGGGTATGAG 199
||||||| |||||||||||||||||||||||||| |||||||||||||||
seq2 CAGGCTCTTGCACACTGACAGCCTACAAGCTGACGCCCAGTGGGTATGAG 200
seq1 TGGGGCCGGCAGAACACAGACAAAGGCAACAACCCCAAAGGCTACCTACC 249
|||| |||||||||||||||||||||||||||||||||||||||||||||
seq2 TGGGTCCGGCAGAACACAGACAAAGGCAACAACCCCAAAGGCTACCTACC 250
seq1 CTCGCACTACGAGAGGGTTCAGATGCTCCTGTCTGACCGATTCCTCGGCT 299
||||||||||||||||||||||||| ||||||||||||||||||||||||
seq2 CTCGCACTACGAGAGGGTTCAGATGATCCTGTCTGACCGATTCCTCGGCT 300
seq1 TCTTCATGGTCCCCGCCCAGTCCTCCTGGAACTACAACTTTATGGGTACG 349
||||||||||||||| |||||||||||||||||||||||||||||||||
seq2 TCTTCATGGTCCCCGAACAGTCCTCCTGGAACTACAACTTTATGGGTACG 350
seq1 TGGTTGTGTGTGCCTAGGAAGTGTGGGGTAGAGACAGGTGACCAGCATGC 399
||||||||||||||||||||||||||||||||||||||||||||||||||
seq2 TGGTTGTGTGTGCCTAGGAAGTGTGGGGTAGAGACAGGTGACCAGCATGC 400
seq1 TTGGTCATCTTGTCATCTTGAGGTCTGGCTGCCTTGGAAACGGTCCAGGG 449
||||||||||||||||||||||||||||||||||||||||||| ||||||
seq2 TTGGTCATCTTGTCATCTTGAGGTCTGGCTGCCTTGGAAACGGCCCAGGG 450
seq1 GCCCAGGCTGGGTGAGGCAGGCAGAACCTTATTAACTTCTGATGTTTCCC 499
|||||||||||||||| |||||||||||||||||||||||||||||||||
seq2 GCCCAGGCTGGGTGAGACAGGCAGAACCTTATTAACTTCTGATGTTTCCC 500
seq1 CTCCCATTAGGTGT-TCGACATGATCCCAACATGAAATACGAGCTGCAGC 548
|||||||||||||| ||||||||||||||||||||||||||||||||||
seq2 CTCCCATTAGGTGTGGCGACATGATCCCAACATGAAATACGAGCTGCAGC 550
seq1 TGGCAAACCCCAAGGAATT 567
|||||||||||||||||||
seq2 TGGCAAACCCCAAGGAATT 569
|
|